Forschungsthemen des Arbeitsgruppe Söding
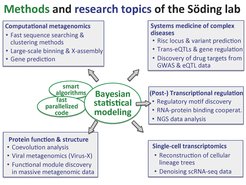
Hochdurchsatzmethoden verãndern die biologische Forschung
Der schnelle Fortschritt besonders in der Hochdurchsatz-Sequenzierung verändert viele Bereiche der Lebenswissenschaften: Nach dem klassischen Forschungsansatz stellen Wissenschaftler Hypothesen auf, die sie anschließend experimentell überprüfen. Mit Hochdurchsatz-Technologien ist stattdessen in bisher unerreichtem Umfang datenbasierte Forschung möglich, die ohne die Voreingenommenheit einer Hypothese auskommt.
Ein Problem von Hochdurchsatz-Methoden ist allerdings, dass die gewonnenen Daten oft ein wesentlich stärkeres »Hintergrundrauschen« enthalten als konventionelle Methoden. Dies limitiert den Nutzen der Messungen oft sehr stark. Unsere Arbeitsgruppe entwickelt daher statistische und bioinformatische Methoden, die präzise und unvoreingenommene Informationen aus Hochdurchsatz-Daten extrahieren können. Unser Ziel ist es, auf diese Weise Daten-basierte Forschungsansätze in der Zell- und Entwicklungsbiologie, Genetik, Mikrobiologie und Systemmedizin zu verbessern.
Sequenzsuchen ermöglichen die Vorhersage von Funktion und Struktur von Proteinen
Wir entwickeln bioinformatische Methoden, um anhand der Aminosäuresequenz von Proteinen ihre räumliche Struktur, Funktion und Evolution vorherzusagen. Mit eigens erarbeiteten statistischen Methoden können wir die immer schneller verfügbaren, riesigen Mengen an Sequenzinformationen nutzen. So wollen wir Lebenswissenschaftlern fortlaufend bessere Werkzeuge zur Verfügung stellen. Unserer Webserver HHpred und die Software HH-suite sind in ihrer Empfindlichkeit führend und vielfach verwendet. HHpred kann nur anhand der Aminosäuresequenz die Verwandtschaft von Proteinen erkennen, die 3 Milliareden Jahre Evolution trennt.

Die Hochdurchsatzseqeunzierung revolutioniert ebenfalls die Mikrobiologie. Es ist heute möglich, Proben aus der Umwelt, z.B. aus unserem Darm, aus dem Meer oder aus Erde zu entnehmen und die genomische DNA und RNA der dort lebenden Mikroorganismen direkt zu sequenzieren. Dies eröffnet vielfältigstw neue Möglichkeiten, die zentralen Rollen der Bakterien, Archäen und Viren zu verstehen, die sie für unsere Gesundheit und unsere Umwelt spielen. Aber den enormen Mengen an Sequenzdaten sind derzeitige Softwaretools nicht gewachsen. Wir entwickeln daher die Sequenzsuchsoftware MMseqs, die extrem schnell und doch sehr empfindlich ist. Weiter entwickeln wir Algorithmen zur extrem schnellen Sequenzclusterung und zur Assemblierung von Genomen aus metagenomischen Sequenzierreads.
Siehe: Quantum leap in fast and deep protein sequence similarity searching
Wie ist die Regulation der Gentranskription in der DNA codiert?
Zweitens wollen wir verstehen, wie in bestimmten Genabschnitten die Regulation der Transkription (der Abschrift der Gene) kodiert ist, der wichtigste zelluläre Regulationsschritt. Um diese Abschnitte zu analysieren und regulatorische Sequenzmotive zu entdecken, entwickeln wir bioinformatische Methoden. Außerdem nutzen wir verschiedene Techniken, um Transkriptionsraten vorherzusagen. Wir arbeiten eng mit experimentellen Laboren zusammen, um herauszufinden, wie die verschiedenen Schritte der Transkription molekular reguliert sind.
Netzwerken von Genen auf der Spur
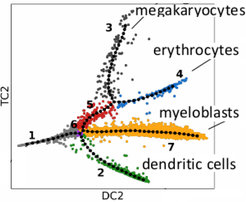
Neuerdings ist es möglich, die Expressionsstärke aller Gene einzelner Zellen zu messen. Die Daten sind stark verrauscht, bieten aber ungeheure neue Möglichkeiten, um zu verstehen, wie zum Beispiel aus einer einzelnen befruchteten Eizelle ein komplexer Organismus entsteht. Die Differentiation von Zellen in spezialisiertere Zelllinien wird kontrolliert durch genregulatorische Netzwerke, die sich durch mathematische Modelle beschreiben lassen. Diesen genregulatorischen Netzwerken können wir durch Messung der Genexpression tausender sich entwickelnder Zellen auf den Grund kommen. Dafuer entwickeln wir statistische Methoden, die auch bei dem starken Rauschen in den Einzelzellmessungen genaue Modelle der zeitlichen Entwicklung der Genexpression ableiten können.
Systemmedizin komplexer Erkrankungen

Schließlich entwickeln wir statistische Methoden, um vorherzusagen, wie es zur Enstehung der häufigsten Erkrankungen wie koronarer Herzkrankheit, Parkinson oder Alzheimer kommt. Wir analysieren grosse Mengen an Genotypdaten kranker und gesunder Patienten und verknüpfen diese mit riesigen Datensätzen zur Messung des Einflusses von Variationen der DNA auf die Expression der Gene. Mithilfe statistischer Modelle können wir dann Gruppen von Genen finden, deren Fehlregulation das Risiko, an bestimmten Krankheiten zu erkranken, erhoehgt.



