Proteinstruktursuche auf neuem Niveau
Dank der methodischen Fortschritte bei der Vorhersage von Proteinstrukturen wie AlphaFold2 und ESMFold nimmt die Anzahl öffentlich zugänglicher Proteinstrukturen rasch zu. Das Wachstum entsprechender Datenbanken ist beispiellos. Strukturen sind stärker konserviert als Proteinsequenzen. Daher können entfernte evolutionäre Beziehungen zwischen Strukturen viel besser nachgewiesen werden als zwischen Proteinsequenzen. Die riesigen Strukturdatenbanken verbessern erheblich die Fähigkeit der Forschenden, entfernt verwandte Proteine zu finden. Somit können sie mehr über die Funktionen von Proteinen anhand bereits erforschter verwandter Moleküle lernen.
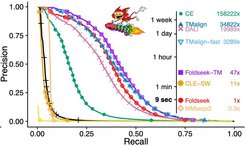
Um diesen Schatz an Strukturdaten effizient zu nutzen, müssen die Datenbanken häufig nach Strukturen durchsucht werden, die den Proteinen von Interesse ähneln. Mit den bestehenden Suchmethoden würde eine einzige Suche unter den 200 Millionen Strukturen in den aktuellen Datenbanken jedoch Wochen oder Monate dauern.
Um diese rechnerische Herausforderung zu bewältigen, haben Forschende der Seoul National University (Südkorea) und des Max-Planck-Instituts für Multidisziplinäre Naturwissenschaften in Göttingen Foldseek entwickelt, ein bahnbrechendes Werkzeug für die Suche nach Proteinstrukturen. Foldseek verkürzt die Suchzeit von Wochen auf nur wenige Sekunden, wobei die Suchempfindlichkeit nur geringfügig unter den empfindlichsten aktuellen Werkzeugen liegt. Um diese hohe Geschwindigkeit zu erreichen, bedient sich Foldseek eines einfachen Tricks: Proteine sind Ketten von chemischen Einheiten, den Aminosäuren, und falten sich zu einer stabilen 3D-Struktur. Foldseek beschreibt diese Struktur als eine Abfolge von Buchstaben, von denen jeder die 3D-Interaktion einer Aminosäure mit der nächstgelegenen Aminosäure kodiert. Foldseek verwendet dann sehr schnelle Sequenzsuchwerkzeuge, um Sequenzen statt 3D-Strukturen zu vergleichen.
Mit seiner überragende Geschwindigkeit und Empfindlichkeit macht Foldseek die neuen, riesigen Datenbanken mit Proteinstrukturen nutzbar, was besonderes in der Molekularbiologie, molekularen Medizin und Mikrobiologie grosse Auswirkungen haben wird. Mit einer Reihe von Merkmalen, Arbeitsabläufen, zusätzlichen Funktionen und einem Webserver (search.foldseek.com) verbessert Foldseek den Prozess der Proteinstruktursuche und der Individualisierung, sodass erfahrene Nutzer*innen ihre Arbeitsabläufe individuell gestalten und Foldseek an ihre Forschungsanforderungen anpassen können. Foldseek erhöht die Geschwindigkeit bei der Suche nach ähnlichen Proteinstrukturen um das Hunderttausendfache, was es zu einem unschätzbaren Werkzeug für zukünftige strukturbasierte Analysen macht. Weitere Informationen finden Sie in der jüngsten Veröffentlichung in Nature Biotechnology.
