Das Ribosom: ein vielseitiges Mega-Ribozym
The ribosome: a versatile mega-ribozyme
Forschungsbericht (importiert) 2013 - Max-Planck-Institut für Multidisziplinäre Naturwissenschaften
Ribosomen – Orte der Proteinsynthese in der Zelle
Proteine sind elementar wichtige Bestandteile von Zellen und Organismen mit vielfältigen Funktionen. Als Katalysatoren sind sie für Stoffwechsel und Energieerzeugung zuständig, als Transporter sorgen sie für den geordneten Stoffaustausch der Zelle mit der Umgebung und für den Stofftransport in der Zelle. Als Strukturproteine sind sie wesentliche Strukturelemente innerhalb und außerhalb der Zellen und als Motorproteine setzen sie Organismen, Zellen und Zellorganellen in Bewegung. Proteine bestehen aus Aminosäuren, deren Reihenfolge Struktur und Eigenschaften der Proteine bestimmt. Die Gesamtheit der Proteine in der Zelle (das "Proteom") umfasst Tausende verschiedener Proteine und wird ständig den wechselnden Bedürfnissen der Zelle angepasst. Im letzten Schritt der Genexpression werden Proteine an Ribosomen synthetisiert. Dabei übersetzen die Ribosomen die genetische Information einer RNA-Kopie (messenger RNA, mRNA) des für ein Protein codierenden Gens in eine Kette von Aminosäuren ("Translation"). Ribosomen sind große (2.5 bis 4 Megadalton, je nach Organismus) Partikel aus zwei verschiedenen Untereinheiten, die aus mehreren, zum Teil sehr großen Molekülen Ribonukleinsäure (ribosomale RNA, rRNA) und zahlreichen Proteinen bestehen.
Entsprechend der zentralen Bedeutung der Proteinsynthese für die Zelle sind Ribosomen evolutiv sehr alt. Die funktionell wichtigen Zentren bestehen aus rRNA und sind hoch konserviert, das heißt in Zusammensetzung und Struktur in allen Organismen sehr ähnlich – entsprechend der Funktion des Ribosoms als Aminosäurepolymerase (Abb. 1). Die Aminosäuren gelangen gebunden an Transfer-RNAs (tRNAs) als Aminoacyl-tRNAs in die A-Stelle des aktiven Zentrums des Ribosoms ("Peptidyltransferase-Zentrum"). Dort reagieren sie mit einer bereits in der P-Stelle gebundenen Peptidyl-tRNA, wobei eine neue Peptidyl-tRNA mit einer um eine Aminosäure verlängerten Peptidkette entsteht. Dieser Vorgang wiederholt sich viele Male, bis das fertige Protein, das aus Hunderten, manchmal auch Tausenden von Aminosäuren bestehen kann, freigesetzt wird. Die Reihenfolge der Aminosäuren im Protein wird durch die Reihenfolge der Bausteine der mRNA festgelegt, die von den Aminoacyl-tRNAs am Ribosom gelesen wird.
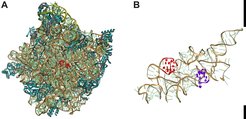
Abb. 1: (A) Die große Untereinheit des Ribosoms aus E. coli. Ribosomale RNAs sind golden (23S rRNA) und gelb (5S rRNA) gefärbt, ribosomale Proteine blau. Das aktive Zentrum ist durch einen gebundenen Inhibitor (rot) gekennzeichnet. (B) Die pseudosymmetrische Struktur der ribosomalen RNA (23S rRNA) im aktiven Zentrum. Die Bindestellen für die tRNA-Substrate in der sog. P-Stelle (rot) und A-Stelle (blau) sind hervorgehoben.
RNA-Katalyse im aktiven Zentrum des Ribosoms
Das Peptidyltransferase-Zentrum (A-Stelle) befindet sich auf der großen Untereinheit der Ribosomen und wird durch eine hoch konservierte Region der rRNA (23S rRNA in Bakterien; 28S rRNA in Eukaryonten) gebildet (Abb. 1A). Die ursprüngliche, primordiale Form der rRNA war vermutlich sehr viel kleiner; durch Verdopplung entstand zunächst eine pseudosymmetrische Struktur, die für die Bindung zweier ähnlicher Substratmoleküle geeignet war und sich im modernen Ribosom mit der um ein Vielfaches größeren rRNA noch erkennen lässt (Abb. 1B). Die Struktur des aktiven Zentrums erlaubt die Bindung der beiden tRNA-Substrate in einer Orientierung, welche die Reaktion zwischen der Aminoacyl-tRNA und der Peptidyl-tRNA begünstigt. Verglichen mit der Reaktion zwischen Modellsubstraten in Lösung ist die Peptidverknüpfung am Ribosom etwa 10 Millionen Mal schneller. Die Frage, welche katalytischen Mechanismen zur Beschleunigung der Reaktion am Ribosom führen, wurde eingehend untersucht [1]. Dabei war ein wichtiger Befund, dass die Katalyse offenbar ohne direkte Beteiligung ribosomaler Gruppen zustande kommt (entropische Katalyse) [2]. Dies machte einen aufgrund älterer Daten vielfach vermuteten Beitrag einer Säure-Basen-Katalyse durch ribosomale Gruppen unwahrscheinlich [3]. Ein solcher Beitrag konnte auch experimentell ausgeschlossen werden [4; 5; 6]. Im Ergebnis kommt die Katalyse der Peptidverknüpfung wohl vor allem dadurch zustande, dass das aktive Zentrum des Ribosoms den Übergangszustand der Reaktion stabilisiert, indem es ein Netzwerk elektrostatischer Wechselwirkungen und Wasserstoffbrücken ausbildet, welches die gleichzeitige Übertragung von drei Protonen in einem zyklischen Übergangszustand begünstigt [7].
Das flexible katalytische Zentrum des Ribosoms
Eine bemerkenswerte Eigenschaft des aktiven Zentrums des Ribosoms ist, dass es neben der Ausbildung von Peptidbindungen zwischen natürlichen Aminosäuren auch den Einbau unnatürlicher Aminosäuren und darüber hinaus noch andere, chemisch zum Teil sehr verschiedene Reaktionen katalysieren kann, etwa die Bildung von Thioestern, Thioamiden oder Phosphinamiden. Physiologisch von Bedeutung ist die hydrolytische Abspaltung des Peptids von Peptidyl-tRNA während der Termination am Ende der Synthese eines Proteins. Das aktive Zentrum wird dabei durch die Bindung eines Terminationsfaktors umprogrammiert, sodass ein Wassermolekül eindringen und die Esterbindung in Peptidyl-tRNA hydrolysieren kann. Im Übergangszustand der Hydrolyse wird nur ein Proton übertragen [7]. Demnach können im aktiven Zentrum des Ribosoms unterschiedlich strukturierte Übergangszustände stabilisiert werden – eine bemerkenswerte Vielseitigkeit oder, anders ausgedrückt, geringe Selektivität. Dies unterscheidet das Ribosom von den üblichen Proteinenzymen, die sich generell durch eine hohe Substratselektivität auszeichnen. Vermutlich ist die geringe Selektivität des Ribosoms, und damit die Flexibilität des aktiven Zentrums, darauf zurückzuführen, dass die RNA im aktiven Zentrum den Übergangszustand durch relativ unspezifische Wechselwirkungen stabilisiert und nicht direkt an der Reaktion teilnimmt. Dies könnte auch der Grund dafür sein, dass die im Vergleich zu Proteinenzymen nicht besonders effektive RNA-Katalyse für Peptidverknüpfung und Peptidyl-tRNA-Hydrolyse in der Evolution beibehalten wurde.
Das Ribosom in der synthetischen Biologie
Die Flexibilität des Peptidyltransferase-Zentrums wird biotechnologisch ausgenutzt zur Herstellung von "Designer-Proteinen", ein zunehmend wichtiges Feld der synthetischen Biologie. Dabei werden unnatürliche Aminosäuren mit den gewünschten chemischen Eigenschaften anstelle natürlicher Aminosäuren in Proteine eingebaut. Für diese "orthologe" Translation wurde eine Reihe verschiedener Strategien entwickelt. Das generelle Problem sind niedrige Ausbeuten, die vor allem auf die Fehlerkorrektur bei der Substratselektion am Ribosom zurückzuführen sind. Um diese Korrekturschritte umgehen zu können, müssen sie im Detail bekannt sein. Die kinetische Untersuchung hat jetzt einen bisher nicht bekannten Schritt bei der Kontrolle der Aminoacyl-tRNA-Bindung an das Ribosom identifiziert, der in diesem Zusammenhang eine Rolle spielt [8]. Die Berücksichtigung dieses Schrittes bei der Auswahl der in Proteine einzubauenden unnatürlichen Aminosäuren könnte Teil einer Strategie sein, die das Problem niedriger Ausbeuten löst.
Der "Prolin-Faktor" EF-P
In den verschiedenen Phasen der Proteinsynthese – Initiation, Elongation, Termination, Recycling – wird das Ribosom durch Translationsfaktoren gesteuert – mit Ausnahme der Peptidverknüpfung in der Elongationsphase. Diese erfolgt üblicherweise spontan unmittelbar nach Bindung des Aminoacyl-tRNA-Substrats, welche die Reaktion kinetisch limitiert. Es war demnach unerwartet, als beobachtet wurde, dass die Aminosäure Prolin sehr langsam eingebaut wird, insbesondere, wenn mehrere Prolinreste aufeinander folgen [9]. Der Grund ist vermutlich, dass die Ringstruktur des Prolins für die Bildung einer Peptidbindung ungünstig ist. Das überraschende Ergebnis der näheren Untersuchung war, dass der Einbau von Prolin mit normaler Geschwindigkeit erfolgt, wenn ein bis dahin wenig untersuchter Translationsfaktor (EF-P) zugegeben wird [10]. Die Bindung von EF-P verhindert, dass das Ribosom bei der Translation von Sequenzen aufeinanderfolgender Prolin-Reste zum Stillstand kommt. Der Effekt kommt wohl dadurch zustande, dass EF-P unmittelbar neben dem aktiven Zentrum des Ribosoms bindet, nachdem die entladene tRNA die sog. E-Stelle (tRNA exit) verlassen hat und dabei die Orientierung der Prolin tragenden tRNAs so verändert, dass die Peptidverknüpfung erleichtert wird (Abb. 2). Auch in anderen Organismen benötigt der Einbau von Prolin in Polyprolin-Sequenzen einen solchen Hilfsfaktor; in Eukaryonten ist es der Faktor eIF5A, ursprünglich als Initiationsfaktor identifiziert.

Abb. 2: Wirkungsweise des Translationsfaktors EF-P beim Einbau von Prolin. EF-P kann nach Dissoziation der tRNA aus der E-Stelle (E) in die freie E-Stelle unmittelbar neben der Peptidyl-tRNA (P) in der P-Stelle binden und verbessert vermutlich ihre Positionierung für die Reaktion mit der Prolyl-tRNA in der A-Stelle (A). Proline sind als rote Kreise, andere Aminosäuren als schwarze Kreise dargestellt.
Von den mehr als 4.000 Genen in E. coli, die bestimmten Funktionen zugeordnet werden konnten, enthalten fast 300 Gene Sequenzen, in denen drei oder mehr Prolinreste aufeinander folgen oder zwei Prolinreste von einem Glycin gefolgt werden. Unter diesen sind Gene für Stoffwechselenzyme, Transporter und regulatorische Transkriptionsfaktoren überrepräsentiert, also Gene, deren Produkte für übergeordnete, die Zelle als Ganze betreffende Vorgänge, wichtig sind. Dies erklärt die bisher nicht verstandene Beobachtung, dass EF-P- Mutationen generelle Auswirkungen auf die Bakterien haben, und etwa zu verminderter Virulenz oder Fitness führen. So enthält das Virulenzprotein EspF, das eine wesentliche Rolle bei Infektionen mit enterohämorrhagischen (EHEC) oder enteropathogenen (EPEC) E. coli-Stämmen spielt, mehrere Polyprolin-Sequenzen. Damit gewinnen EF-P und insbesondere die Enzyme, die EF-P durch Einführung einer Modifizierung aktivieren, Interesse als mögliche Zielstrukturen in pathogenen Bakterien. Da sie in Eukaryonten fehlen, könnten sie für die Entwicklung neuer Wirkstoffe genutzt werden.
Ausblick
Das Ribosom ist ein evolutionäres Fossil und die Untersuchung der Funktionen dieser RNA-basierten Nanomaschine führt uns weit zurück in die Zeit der Entstehung des Lebens – und gleichzeitig in die Zukunft der synthetischen Biologie. Das vertiefte Verständnis der enzymatischen Funktion dieses größten bekannten Ribozyms ist dafür unerlässlich. Aber auch bei anderen Funktionen des Ribosoms gibt es noch viele wichtige offene Fragen, zum Beispiel wie thermische Fluktuationen zur Auslösung von gerichteten molekularen Bewegungen ausgenutzt werden oder wie programmierte Abweichungen von der regulären Funktion zustande kommen und zu neuen Produkten der Proteinsynthese führen können.

