Elektronenspins als Sonden in Biomolekülen
Electron spins as probes for biomolecules
Forschungsbericht (importiert) 2009 - Max-Planck-Institut für Multidisziplinäre Naturwissenschaften
Ob einfaches Wasser oder komplizierte Proteine, in Atomen und Molekülen treten Elektronen meist paarweise auf. Durch ihren Eigendrehimpuls – den sogenannten Spin – erzeugen sie ein Magnetfeld, doch wird dieses durch die entgegengesetzte Ausrichtung der gekoppelten Elektronen aufgehoben. Ungepaarte Elektronen dagegen weisen ein nicht verschwindendes magnetisches Moment auf, das sich mithilfe der Elektronen-Spin-Resonanz (ESR oder EPR) detektieren lässt. Dieses Moment – etwa um drei Größenordnungen stärker als das Moment eines Protons – kann als Sonde für hochempfindliche Messungen in picomolaren Spinkonzentrationen dienen, um magnetische Wechselwirkungen in Reichweiten auf der atomaren bis hin zur Nanometerskala zu detektieren.
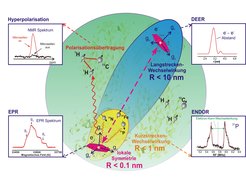
Ungepaarte Elektronen kommen typischerweise in organischen Radikalen oder Übergangsmetallen vor. Solche sogenannten paramagnetischen Zentren sind manchmal in Proteinen oder Nukleinsäuren natürlich enthalten oder können künstlich als Markierungen eingeführt werden. Für strukturelle Untersuchungen mit EPR sind drei Arten von physikalischen Wechselwirkungen von zentraler Bedeutung: a) lokale (Spin-Bahn-) Wechselwirkungen b) magnetische Elektron-Kern-Wechselwirkungen und c) Elektron-Elektron-Wechselwirkungen. Entsprechend gehört zur modernen gepulsten EPR-Spektroskopie ein breites Repertoire an Methoden, um diese Wechselwirkungen selektiv zu detektieren (Abb. 1).
Die Forschungsgruppe am MPI für biophysikalische Chemie hat in ihrem Labor Techniken entwickelt, um paramagnetische Zentren gleichzeitig mit Mikrowellen oder Radiofrequenzstrahlung anzuregen und ihre magnetischen Wechselwirkungen zu manipulieren. So wurden Abstände zwischen den aktiven Zentren eines Proteins bis in den Nanometerbereich vermessen und die Ausrichtung dieser aktiven Zentren innerhalb des Proteins bestimmt. Der Einsatz sehr unterschiedlicher Anregungsfrequenzen (Wellenlänge von cm bis mm) ermöglicht weiterhin, die gewünschten Interaktionen gezielt nach deren Größe zu selektieren. Anregungsfrequenzen im Millimeter-Bereich erfordern nicht nur supraleitende polarisierende Magnetfelder, sondern auch eine deutlich komplexere Technik für die im Nanosekundenbereich stattfindende Signalanregung und Detektion. Diese biophysikalischen Untersuchungen gehen daher Hand in Hand mit methodischen und technischen Weiterentwicklungen.
Abstandsmessungen in hohen Magnetfeldern
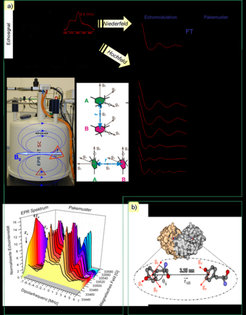
Abstandsmessungen mit EPR, spezifisch mit Elektron-Elektron-Doppelresonanz (DEER), beruhen auf der Bestimmung der dipolaren Wechselwirkung zwischen zwei paramagnetischen Zentren, die nicht nur vom Abstand, sondern auch von der Ausrichtung des Abstandsvektors zum externen Magnetfeld abhängt. Mithilfe der Hochfeld-EPR-Spektroskopie können Orientierungen einzelner Moleküle im EPR-Spektrum aufgelöst werden, sodass ein neuer Parameter (die Molekül-Orientierung) erfasst werden kann (dargestellt in Abb. 2). Eine quantitative Analyse dieses dreidimensionalen Diagramms lässt Rückschlüsse auf die gegenseitige Orientierung der paramagnetischen Zentren zu. Mittels dieser Methode konnte beispielsweise die Ausrichtung der essenziellen Tyrosylradikale im aktiven Komplex der Ribonukleotid-Reduktase (RNR) bestimmt werden, die wichtige Informationen über die Struktur des Proteinkomplexes lieferte.
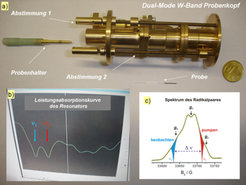
Eine der größten Herausforderungen solcher Abstandsmessungen in hohen Feldern stellt die Konstruktion des Mikrowellenresonators dar, in dem die Mikrowellenstrahlung verstärkt und auf die Probe gestrahlt wird. Die Messmethode erfordert zwei Mikrowellenstrahlen unterschiedlicher Frequenz, um zwei gekoppelte Zentren jeweils selektiv anzuregen und zu detektieren. Für diesen Zweck wurde ein neuartiger Resonator entwickelt, der zwei Mikrowellenfrequenzen in vergleichbarem Faktor verstärken kann. Der Resonator mit seiner charakteristischen Leistungsabsorptionskurve ist in Abbildung 3 dargestellt.
Studien von enzymatischen Reaktionen
In biologischen Systemen verlaufen zahlreiche Prozesse über paramagnetische Intermediate, die durch Redoxreaktionen oder Radikal- bzw. Elektronentransfer-Mechanismen entstehen. Ein Paradebeispiel dafür ist das Enzym Ribonukleotid-Reduktase, das in allen lebenden Organismen die Reduktion von Nukleotiden zu Desoxynukleotiden katalysiert. Mithilfe verschiedener EPR-Methoden, insbesondere der Hochfeld-EPR-Spektroskopie, konnten díe Forscher über das letzte Jahrzehnt mehrere enzymatische Zwischenschritte aufklären. Zur Zeit untersuchen sie den Mechanismus des langreichweitigen (über 35 Å) Elektronentransfers zwischen den Enzymuntereinheiten und konnten bereits erfolgreich drei Aminosäureradikale als Intermediate identifizieren (dargestellt in Abb. 4). Die Kombination von EPR- und Elektron-Kern-Doppelresonanz (ENDOR)-Techniken erlaubte weiterhin, den postulierten Protonen-gekoppelten Elektronentransfer-Mechanismus zu untersuchen. An den essentiellen Tyrosinen Y356, Y730 und Y731 wurden Wasserstoffbrücken identifiziert, für die auch aufgrund theoretischer Berechnungen eine wichtige Rolle am Elektronentransfer vorgeschlagen wurde.
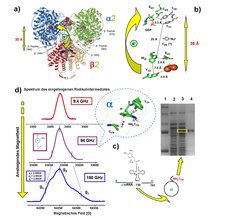
Ein zweites Beispiel für einen Enzymprototypen ist die Heterodisulfid-Reduktase (HDR) aus methanogenen Archaea. Sie verwendet einen neuartigen paramagnetischen Eisen-Schwefel-Metallcluster als katalytisches Zentrum. Das Bindungsmotiv dieses aktiven Zentrums wurde in einer Proteinfamilie mit mehr als 1800 Mitgliedern gefunden; doch nur bei der HDR ist die Funktion dieses Zentrums bekannt. Mithilfe der EPR/ENDOR-Spektroskopie konnte die Forschungsgruppe die Struktur dieses Clusters sowie die Bindungsstelle des Substrats an den Metallcluster nachweisen.
Studien von Nukleinsäuren oder diamagnetischen Proteinen mittels Spinmarkierungen
EPR kann in einem viel breiteren Umfang auf diamagnetische Proteine oder makromolekulare Komplexe angewandt werden, wenn man Spinmarkierungen selektiv und kontrolliert einführen kann. Mit dieser Technik untersuchen die Wissenschaftler sowohl lokale dynamische Eigenschaften als auch globale strukturelle Eigenschaften von Protein-Komplexen mittels Abstandsmessungen im Nanometerbereich. Zu den attraktiven Aspekten dieser Methodik zählen die hohe Spinempfindlichkeit (die geringfügige Probenmengen von ≤1 µl bei 94 GHz erfordert), die geringe Molekülgröße der Markierungen (je nach Art zwischen etwa 4 und 8 Å) und schließlich die Unabhängigkeit der Messung von der Proteingröße.
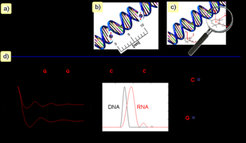
Zu den stabilen organischen Radikalen, die für Spinmarkierungen eingesetzt werden, zählen vor allem die sogenannten Nitroxide (gezeigt in Abb. 5). Das chemische Verfahren zum Einführen dieser Markierungen hat in der Vergangenheit große Herausforderungen an Wissenschaftler gestellt. Mittlerweile konnten jedoch mehrere Protokolle entwickelt werden, um Spinmarkierungen sowohl in Nukleinsäuren als auch in Proteine einzuführen. In Abb. 5 ist eine repräsentative Abstandsmessung an markierten DNA- und RNA-Duplexen dargestellt. Die beobachtete breitere Abstandsverteilung in der RNA-Probe spiegelt die höhere Beweglichkeit und Konformationsdynamik von RNA im Vergleich zu DNA wider. Mit diesem Messprotokoll sollen in Zukunft RNA-basierte enzymatische „Maschinen“ untersucht werden, die wichtige zelluläre Funktionen übernehmen.




