Strukturbestimmung von supramolekularen Proteinanordnungen mit Hilfe von Festkörper-NMR-Spektroskopie
Forschungsbericht (importiert) 2013 - Max-Planck-Institut für Multidisziplinäre Naturwissenschaften
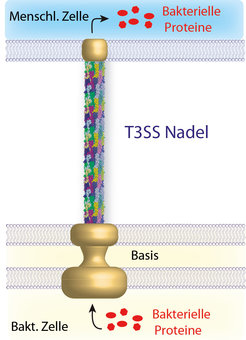
Abb. 1: Schematische Darstellung des bakteriellen Typ-III-Sekretionssystems. Die Struktur der Nadel wurde kürzlich mittels einer Kombination aus Festkörper-NMR, Elektronenmikroskopie und Computermodellierung bestimmt. Durch die hohle Nadel werden Proteine aus der bakteriellen Zelle in die menschliche Wirtszelle eingeschleust.
Trotz beeindruckender Fortschritte in der Strukturbiologie gibt es immer noch einige Klassen von Proteinen bzw. Proteinkomplexen, die sich einer strukturellen und dynamischen Beschreibung durch Standardmethoden wie der Röntgenkristallographie oder der Lösungs-NMR-Spektroskopie widersetzen. Dazu gehören z. B. Membranproteine in ihrer natürlichen Lipidumgebung, Amyloidfibrillen, die eine wichtige Rolle in neurodegenerativen Erkrankungen spielen, oder auch große supramolekulare Proteinkomplexe. Solche Systeme lassen sich in der Regel nur schwer kristallisieren und ihre Unlöslichkeit verhindert die Anwendung von Lösungs-NMR-Spektroskopie. Mit Festkörper-NMR-Spektroskopie lassen sich dagegen Proteine untersuchen, die weder eine hohe Ordnung wie in einem Kristall aufweisen noch löslich sind. Dazu werden die zu untersuchenden Proben in starke supraleitende Magneten gebracht (externe Felder bis 20 Tesla, d.h. ~ 400000-mal so stark wie das Erdmagnetfeld) und in schnelle Rotation versetzt (ca. 10000 Umdrehungen pro Sekunde). Diese Rotation um eine Achse, die zu dem Magnetfeld um den „magischen“ Winkel von 54.7° gekippt ist, simuliert die Situation von schnell und frei taumelnden Molekülen in Lösung. Mit Hilfe dieses sogenannten magic-angle-spinning erreicht man eine hohe Auflösung und Sensitivität in den NMR-Spektren von Proteinen in fester Phase. Dabei handelt es sich bei den festen Proteinproben in der Regel nicht um ein staubtrockenes Pulver, sondern eher um ein hochviskoses Material wie beispielsweise pelletierte Proteoliposomen oder abzentrifugierte Amyloidfibrillen.
Mit Hilfe von magic-angle-spinning Festkörper-NMR-Spektroskopie wurden in den letzten Jahren eine Reihe von Membranproteinen untersucht, z. B. der spannungsabhängige Anionenkanal (VDAC) [2]. VDAC ist ein Protein der äußeren Mitochondrienmembran und stellt dort den wichtigsten Transportweg für ADP, ATP und andere Metaboliten dar. Im Vergleich zur Röntgenkristallographie, die in der Strukturbestimmung von Membranproteinen exzellente Fortschritte erzielt hat – als ein herausragendes Beispiel seien hier die G-Protein gekoppelten Rezeptoren genannt – hat die Festkörper-NMR den Vorteil, dass die Proteine in einer physiologischen Lipidumgebung bei Raumtemperatur studiert werden können. Weiterhin eignet sich die NMR-Spektroskopie im Allgemeinen hervorragend, um neben der Struktur auch die Proteindynamik zu charakterisieren, welche oft in einem unmittelbaren Zusammenhang mit der Proteinfunktion steht. Eine weitere wichtige Anwendung von Festkörper-NMR stellt die Strukturbestimmung von Amyloidfibrillen dar. Dabei können einerseits die Sekundärstruktur des Proteins bestimmt und weiterhin intra- und intermolekulare Abstände in den Aggregaten gemessen werden. So konnte beispielsweise gezeigt werden, dass Amyloidfibrillen, gebildet aus den Proteinen Tau [3] und α-Synuklein [4], welche eine Rolle in neurodegenerativen Erkrankungen wie Alzheimer und Parkinson spielen, eine parallele beta-Faltblatt-Architektur aufweisen.
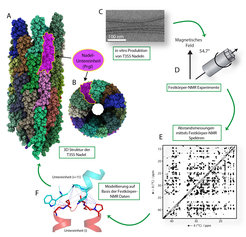
Abb. 2: Eine neue Methode, um die Struktur der Nadel des Typ-3-Sekretionssystems (T3SS) und ähnlicher supramolekularer Anordnungen zu bestimmen. (A) Seitenansicht und (B) Blick von oben auf die Festkörper-NMR-Struktur der Nadel von S. typhimurium. (C) Elektronen-mikroskopische Aufnahme von in vitro (im Labor) hergestellten T3SS-Nadeln. (D) Festkörper-NMR-Rotor und (E) 13C-13C-Korrelationsspektrum. (F) Intermolekulare Kontakte (in blau gestrichelt) zwischen zwei PrgI-Untereinheiten in der Nadelanordnung.
Besonders gut geeignet für die Festkörper-NMR sind homo-oligomere supramolekulare Proteinanordnungen wie die Nadel des Typ-III-Sekretionssystems (T3SS). Das T3SS wird von zahlreichen gram-negativen Bakterien verwendet, um Effektorproteine in Wirtszellen einzuschleusen. Dort manipulieren die Substanzen wichtige Stoffwechselvorgänge und setzen die Immunabwehr der infizierten Zellen außer Gefecht – mit fatalen Folgen. Der Erreger kann sich nun ungehindert im Organismus ausbreiten. Eine schematische Darstellung des T3SS ist in Abbildung 1 gegeben. Die Basis des T3SS ist fest in der inneren und äußeren bakteriellen Membran verankert. Ein Kanal durch die Basis mündet dann in die hohle extrazelluläre Nadel. Diese wird aus etwa 100-150 Kopien eines einzelnen kleinen Proteins gebildet – im Falle von Salmonella typhimurium dem 80-Reste-Protein PrgI. Kürzlich konnte gezeigt werden, dass die Nadeln des T3SS auch im Labor nachgebaut werden können – durch Selbstassoziation des Proteins PrgI (siehe Abb. 2C) – und dass Festkörper-NMR Spektren dieser In-vitro-Nadeln eine außerordentlich hohe spektrale Auflösung aufweisen [6]. Das deutet darauf hin, dass die PrgI-Untereinheiten in den Nadeln extrem gleichmäßig gepackt sind, quasi wie in einem Kristall.
In Folge der ausgezeichneten Datenqualität konnten die Wissenschaftler um Adam Lange eine atomare Struktur der Nadel bestimmen. Dazu wurden Festkörper-NMR-Daten mit Elektronmikroskopie und Computermodellierung kombiniert. Die so bestimmte Struktur [6] repräsentiert eine der komplexesten und größten Proteinstrukturen, die bisher mit Festkörper-NMR bestimmt werden konnten. Die Nadelstruktur zeigt, dass der Innendurchmesser der Nadel nur etwa zwei Nanometer beträgt. Die sekretierten Effektorproteine müssen daher die Nadel im entfalteten Zustand passieren und sich in der Wirtszelle zunächst einmal wieder richtig falten, bevor sie ihre Wirkung entfesseln können. In etablierten Modellen der T3SS-Nadel auf Basis von niedrig aufgelösten elektronenmikroskopischen Messungen war bisher angenommen worden, dass der zwischen verschiedenen Spezies konservierte C-Terminus an der äußeren Oberfläche der Nadel lokalisiert ist. Dagegen zeigt die Festkörper-NMR-Struktur, dass der C-Terminus tatsächlich im Innern der Nadel liegt und die weniger konservierten Bereiche im N-Terminus des Nadelproteins an der Oberfläche. Diese Wandelbarkeit der Oberfläche könnte eine Strategie der Bakterien sein, um der Immunabwehr des Wirts zu entkommen. Veränderungen auf der Nadeloberfläche könnten es dem Immunsystem des Wirts dann erschweren, den Erreger wiederzuerkennen.
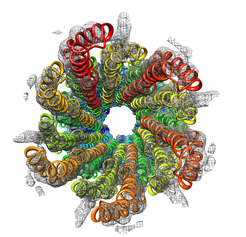
Abb. 3: Blick durch die T3SS-Nadel. Die schematische Darstellung der PrgI-Untereinheiten entspricht der Festkörper-NMR-Struktur. Das graue Netzwerk im Hintergrund repräsentiert die Elektronendichte aus der kryoelektronenmikroskopischen Untersuchung von Fujii et al. [7].
Kürzlich gelang es in einer elektronenmikroskopischen Studie, Daten der Shigella flexneri-Nadel in höherer Auflösung als bisher aufzunehmen [7]. Ein Vergleich von Festkörper-NMR-Daten an Nadeln in Salmonella typhimurium und Shigella flexneri zeigte, dass die Nadeln beider Spezies eine gemeinsame Architektur aufweisen [8]. Auf dieser Basis konnte ein Strukturmodell für die Shigella-Nadel vorgeschlagen werden und mit den elektronenmikroskopischen Daten von Fujii et al. verglichen werden (siehe Abb. 3). Der Vergleich zeigt, dass die unabhängig bestimmte Elektronendichte sehr gut durch das Festkörper-NMR-Modell erklärt wird. Kürzlich konnte des Weiteren eine höher aufgelöste Festkörper-NMR-Struktur eines PrgI-Tetramers gelöst werden, die es erlaubt, die Händigkeit der Nadel festzulegen [9]. Dieser Parameter war in der vorherigen Studie nicht eindeutig bestimmt worden [6]. Die T3SS-Nadel besitzt eine rechtshändige helikale Struktur mit etwa 11 PrgI-Untereinheiten in zwei helikalen Umdrehungen und ähnelt darin sehr den verwandten Flagellen.
Da durch elektronenmikroskopische Untersuchungen erlangte Elektrondichten Informationen über die globale Form des untersuchten Proteinkomplexes liefern und Festkörper-NMR-Daten im Gegensatz dazu lokale strukturelle Parameter (dihedrale Winkel des Peptidrückgrats, d.h. Sekundärstruktur, und intra- und intermolekulare Abstände zwischen 13C- und/oder 15N-Kernen, d.h. Tertiär- und Quartärstruktur), ergänzen sich beide Methoden ideal. In Zukunft kann also erwartet werden, dass weitere Hybridstrukturen von supramolekularen Proteinanordnungen gelöst werden, bei denen Informationen, gewonnen durch (Kryo)-Elektronenmikroskopie und Festkörper-NMR, und möglicherweise auch Kristallstrukturen bzw. NMR-Strukturen von monomeren Untereinheiten mit einfließen.


![Abb. 3: Blick durch die T3SS-Nadel. Die schematische Darstellung der PrgI-Untereinheiten entspricht der Festkörper-NMR-Struktur. Das graue Netzwerk im Hintergrund repräsentiert die Elektronendichte aus der kryoelektronenmikroskopischen Untersuchung von Fujii et al. [7]. Abb. 3: Blick durch die T3SS-Nadel. Die schematische Darstellung der PrgI-Untereinheiten entspricht der Festkörper-NMR-Struktur. Das graue Netzwerk im](https://www.mpg.de/11604967/original-1508157179.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MDQ5Njd9--3aeba496fa4be063013a7979b5b995fe7fb6f3fc)