Untersuchung an molekularen Komplexen mittels hochauflösender Festkörper-NMR-Spektroskopie
Forschungsbericht (importiert) 2007 - Max-Planck-Institut für Multidisziplinäre Naturwissenschaften
Einleitung
In den letzten Jahren wurden bedeutende Fortschritte erzielt, um mittels hochauflösender Festkörper-NMR-Methoden heterogene Systeme wie Proteoliposomen, Mikrokristalle und präzipitierte oder fibrillisierte Moleküle auf struktureller Basis zu untersuchen. Dabei wird die Wechselwirkung von magnetischen Momenten, die bei einer Vielzahl von Atomen vorhanden sind, mit einem äußeren magnetischen Feld mit spektroskopischen Methoden untersucht. Strukturelle Information gewinnt man dabei unter anderem aus der NMR-Resonanzfrequenz einer einzelnen Atomposition, welche oft direkt von der molekularen Struktur abhängt, und der Messung von interatomaren Abständen. Spektroskopisch werden diese Wechselwirkungen dann im Rahmen von mehrdimensionaler Korrelationspektroskopie für viele verschiedene Atome des zu untersuchenden Systems analysiert [1].
Anwendungen
(1) Proteinaggregation
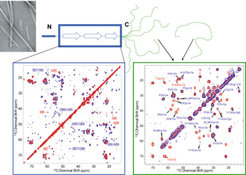
Amyloidose beschreibt einen Faltungsprozess, bei dem Proteine keine wohldefinierte Struktur einnehmen und tritt bei der Typ II-Diabetes sowie der Alzheimer- und Parkinson-Krankheit auf. Da Proteinfibrillen, sofern sie nicht aus kurzen Peptiden bestehen, nicht kristallin und schwer löslich sind, hat sich die Festkörper-NMR zu der führenden Technik zur strukturellen Analyse entwickelt. In Zusammenarbeit mit Kollegen am MPI für biophysikalische Chemie wurde in der AG Festkörper-NMR die Fibrillenbildung von α-Synuclein untersucht, welches aus 140 Aminosäuren besteht und eine große Rolle bei der Parkinson'schen Krankheit spielt. Dazu wurden neue Festkörper-NMR-Methoden entwickelt [2, 3], die eine spektrale Trennung der festen Kernstruktur der α-Synuclein-Fibrillen sowie der schnell beweglichen C-terminalen Enden bzw. der löslichen Monomere erlauben (Abb. 1). Die Festkörper-NMR-Messungen führten zu dem Ergebnis, dass α-Synuclein-Fibrillen aus einer zentralen Region bestehen, welche reich an β-Faltblatt-Strukturen ist, sowie einem hoch flexiblen C-Terminus und einem ungeordneten N-Terminus. In Zusammenarbeit mit anderen Forschungsgruppen innerhalb der Max-Planck-Gesellschaft werden z.Zt. weitere Proteine, die im Zusammenhang mit neurodegenerativen Erkrankungen eine Rolle spielen, mittels Festkörper-NMR-Methoden untersucht.
(2) Membranproteine
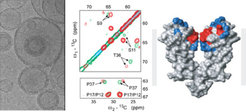
Das Studium der Struktur und Dynamik von Membranprotein steht in engem Zusammenhang mit dem Verständnis elementarer biologischer Prozesse wie Proteinsynthese, Transport und Signaltransduktion. Ionenkanäle steuern z.B. den selektiven Transport von Ionen in einem elektrochemischen Gradienten und werden durch äußere Moleküle (sog. Liganden) beeinflusst. Eine solche Wechselwirkung, d.h. die Bindung eines Toxins an einen Kaliumkanal, wurde mittels Festkörper-NMR in künstlichen Zellmembranen untersucht. In einem ersten Satz von Experimenten wurde dazu die Struktur des Toxins mittels Festkörper-NMR bestimmt [4]. Anschließend wurde durch Vergleichsexperimente des Ionenkanals in An- und Abwesenheit des Toxins das Bindungsverhalten auf atomarer Ebene analysiert [5]. Dabei zeigte es sich, dass sowohl Ligand als auch Ionenkanal ihre Struktur bei der Komplexbildung ändern (Abb. 2). Diese Beobachtung liefert neue Einblicke in die Funktionalität einer wichtigen Klasse von Membranproteinen und könnte bei der pharmakologischen Suche von effizienteren Medikamenten von Nutzen sein.
Ausblick
Die Festkörper-NMR hat in den letzten Jahren beachtliche Fortschritte bei der Analyse von biologischen Systemen gemacht. Weitere Verbesserungen im Bereich der Methodenentwicklung werden es ermöglichen, Biomoleküle von zunehmender Größe und Komplexität zu untersuchen. Die Kombination solcher Ansätze mit anderen biophysikalischen Techniken wird es zukünftig erlauben, elementare biologische Prozesse umfassend zu verstehen.
Danksagung
Die hier beschriebenen Resultate wurden in der AG Festkörper-NMR, z.T. in wissenschaftlicher Zusammenarbeit mit Kollegen in und außerhalb des MPIs für biophysikalische Chemie, erzielt.

